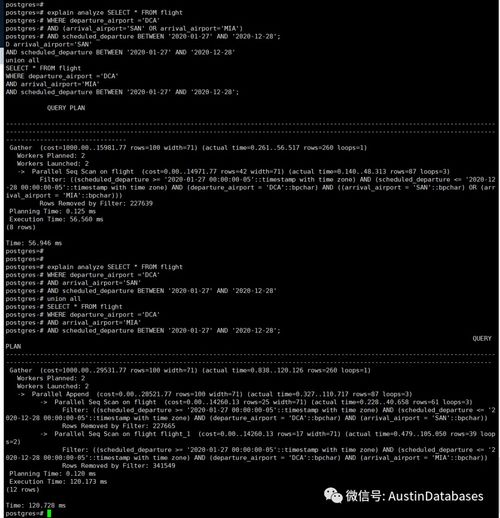
高斯朴素贝叶斯 是一种基于概率方法和高斯分布的机器学习的分类技术。 高斯朴素贝叶斯假设每个参数具有预测输出变量的独立能力。 所有参数的预测组合是最终预测,它返回因变量被分类到每个组中的概率,最后的分类被分配给概率较高的分组。
什么是高斯分布?

高斯分布也称为正态分布,是描述自然界中连续随机变量的统计分布的统计模型。 正态分布由其钟形曲线定义, 正态分布中两个最重要的特征是均值 和标准差 。 平均值是分布的平均值,标准差是分布在平均值周围的“宽度”。
重要的是要知道正态分布的变量 从 -∞ < X < +∞ 连续分布,并且模型曲线下的总面积为 1。
多分类的高斯朴素贝叶斯
导入必要的库:
from random import randomfrom random import randintimport pandas as pdimport numpy as npimport seaborn as snsimport matplotlib.pyplot as pltimport statisticsfrom sklearn.model_selection import train_test_splitfrom sklearn.preprocessing import StandardScalerfrom sklearn.naive_bayes import GaussianNBfrom sklearn.metrics import confusion_matrixfrom mlxtend.plotting import plot_decision_regions
现在创建一个预测变量呈正态分布的数据集。
#Creating values for FeNO with 3 classes:FeNO_0 = np.random.normalFeNO_1 = np.random.normalFeNO_2 = np.random.normal#Creating values for FEV1 with 3 classes:FEV1_0 = np.random.normalFEV1_1 = np.random.normalFEV1_2 = np.random.normal#Creating values for Broncho Dilation with 3 classes:BD_0 = np.random.normalBD_1 = np.random.normalBD_2 = np.random.normal#Creating labels variable with three classes:disease possible disease no disease:not_asthma = np.zeros, dtype=int)poss_asthma = np.ones, dtype=int)asthma = np.full, 2, dtype=int)#Concatenate classes into one variable:FeNO = np.concatenateFEV1 = np.concatenateBD = np.concatenatedx = np.concatenate#Create Dataframe:df = pd.Dataframe#Add variables to Dataframe:df['FeNO'] = FeNO.tolistdf['FEV1'] = FEV1.tolistdf['BD'] = BD.tolistdf['dx'] = dx.tolist#Check database:df
我们的df有 600 行和 4 列。 现在我们可以通过可视化检查变量的分布:
fig, axs = plt.subplots)sns.kdeplotsns.kdeplotsns.kdeplotsns.distplotsns.distplotsns.distplotplt.show
通过人肉的检查,数据似乎接近高斯分布。 还可以使用 qq-plots仔细检查:
from statsmodels.graphics.gofplots import qqplotfrom matplotlib import pyplot#q-q plot:fig, axs = pyplot.subplots)qqplotqqplotqqplotpyplot.show
虽然不是完美的正态分布,但已经很接近了。下面查看的数据集和变量之间的相关性:
#Exploring dataset:sns.pairplotplt.show
可以使用框线图检查这三组的分布,看看哪些特征可以更好的区分出类别
# plotting both distibutions on the same figurefig, axs = plt.subplots)fig = sns.kdeplotfig = sns.kdeplotfig = sns.kdeplotsns.boxplotsns.boxplotsns.boxplotplt.show
手写朴素贝叶斯分类
手写代码并不是让我们重复的制造轮子,而是通过自己编写代码对算法更好的理解。在进行贝叶斯分类之前,先要了解正态分布。

正态分布的数学公式定义了一个观测值出现在某个群体中的概率:
我们可以创建一个函数来计算这个概率:
def normal_dist: prob_density = ) * np.exp/sd)**2) return prob_density
知道正态分布公式,就可以计算该样本在三个分组概率。 首先,需要计算所有预测特征和组的均值和标准差:
#Group 0:group_0 = df[df['dx'] == 0]print)print)print)print)print)print)#Group 1:group_1 = df[df['dx'] == 1]print)print)print)print)print)print)#Group 2:group_2 = df[df['dx'] == 2]print)print)print)print)print)print)
现在,使用一个随机的样本进行测试:FEV1 = 2.75FeNO = 27BD = 125
#Probability for:#FEV1 = 2.75#FeNO = 27#BD = 125#We have the same number of observations, so the general probability is: 0.33Prob_geral = round#Prob FEV1:Prob_FEV1_0 = round, 10)printProb_FEV1_1 = round, 10)printProb_FEV1_2 = round, 10)print#Prob FeNO:Prob_FeNO_0 = round, 10)printProb_FeNO_1 = round, 10)printProb_FeNO_2 = round, 10)print#Prob BD:Prob_BD_0 = round, 10)printProb_BD_1 = round, 10)printProb_BD_2 = round, 10)print#Compute probability:Prob_group_0 = Prob_geral*Prob_FEV1_0*Prob_FeNO_0*Prob_BD_0printProb_group_1 = Prob_geral*Prob_FEV1_1*Prob_FeNO_1*Prob_BD_1printProb_group_2 = Prob_geral*Prob_FEV1_2*Prob_FeNO_2*Prob_BD_2print
可以看到,这个样本具有属于第 2 组的概率最高。这就是朴素贝叶斯手动计算的的流程,但是这种成熟的算法可以使用来自 Scikit-Learn 的更高效的实现。
Scikit-Learn的分类器样例
Scikit-Learn的GaussianNB为我们提供了更加高效的方法,下面我们使用GaussianNB进行完整的分类实例。首先创建 X 和 y 变量,并执行训练和测试拆分:
#Creating X and y:X = df.dropy = df['dx']#Data split into train and test:X_train, X_test, y_train, y_test = train_test_split
在输入之前还需要使用 standardscaler 对数据进行标准化:
sc = StandardScalerX_train = sc.fit_transformX_test = sc.transform
现在构建和评估模型:
#Build the model:classifier = GaussianNBclassifier.fit#evaluate the model:print)print)
下面使用混淆矩阵来可视化结果:
# Predicting the Test set resultsy_pred = classifier.predict#Confusion Matrix:cm = confusion_matrixprint

通过混淆矩阵可以看到,的模型最适合预测类别 0,但类别 1 和 2 的错误率很高。为了查看这个问题,我们使用变量构建决策边界图:
df.to_csvdata = pd.read_csvdef gaussian_nb_a: x = data[['BD','FeNO',]].values y = data['dx'].astype.values Gauss_nb = GaussianNB Gauss_nb.fit print) #Plot decision region: plot_decision_regions #Adding axes annotations: plt.xlabel plt.ylabel plt.title plt.showdef gaussian_nb_b: x = data[['BD','FEV1',]].values y = data['dx'].astype.values Gauss_nb = GaussianNB Gauss_nb.fit print) #Plot decision region: plot_decision_regions #Adding axes annotations: plt.xlabel plt.ylabel plt.title plt.showdef gaussian_nb_c: x = data[['FEV1','FeNO',]].values y = data['dx'].astype.values Gauss_nb = GaussianNB Gauss_nb.fit print) #Plot decision region: plot_decision_regions #Adding axes annotations: plt.xlabel plt.ylabel plt.title plt.showgaussian_nb_agaussian_nb_bgaussian_nb_c
通过决策边界我们可以观察到分类错误的原因,从图中我们看到,很多点都是落在决策边界之外的,如果是实际数据我们需要分析具体原因,但是因为是测试数据所以我们也不需要更多的分析。
https://www.overfit.cn/post/0457f85f2c184ff0864db5256654aef1
作者:Carla Martins
免责声明:本平台仅供信息发布交流之途,请谨慎判断信息真伪。如遇虚假诈骗信息,请立即举报
举报